リボゾームDNAの変異に基づく果樹ハダニ類の系統関係の解析
※アーカイブの成果情報は、発表されてから年数が経っており、情報が古くなっております。
同一分野の研究については、なるべく新しい情報を検索ください。
要約
制限酵素を用いて,ハダニ類のDNAを切断したときに得られたリボゾームDNA(rDNA)断片の長さの変異から,クワオオハダニとその近縁種および他属のナミハダニの類縁関係を推定した。
- 担当:果樹試験場 安芸津支場 虫害研究室
- 連絡先:成果情報のお問い合わせ
背景・ねらい
クワオオハダニは従来ミカンハダニと同種(休眠系統)として取り扱われてきた。当研究室ではミカンハダニの休眠性が異なる系統を電気泳動法を用いて迅速に判別する手法を開発し,この手法を用いて非休眠系統と休眠系統が生物学的に別種であることを明らかにした。これらの結果と大学等で同時に行われていた試験結果に基づいて分類学的に再検討され,休眠系統はミカンハダニとは別種のクワオオハダニとして取り扱われるようになった。そこで,従来よりミカンハダニの近縁種として知られているリンゴハダニを含め,近縁なこれら3種について分子遺伝学的見地から系統関係を検討した。
成果の内容・特徴
- 微量(数百頭)のハダニ類からDNAを抽出・精製する技術を確立した。
- Kominami et al.(1981)により単離されたマウスのrDNA断片が,ハダニ類のrDNA断片の検出に有効であることを明らかにした。
- 神奈川県平塚市,愛媛県松山市および福岡県山川町産のミカンハダニと北海道札幌市および鳥取県大栄町産のクワオオハダニならびに福島県福島市産のリンゴハダニと当支場で累代飼育しているナミハダニから抽出した全DNAを12種類の制限酵素で消化・断片化した後,マウスのrDNA断片(図1)をプローブとしたサザン・ハイブリダイゼーション法により,ハダニ類のrDNA断片を検出した。
- ハダニ類のrDNA断片は全個体群で71本が検出された。また,ハダニ類のrDNAの反復単位は10kbp 前後と推定され,この長さはミカンハダニとクワオオハダニでほぼ同等で,リンゴハダニではこれらよりやや長く,ナミハダニではやや短かった。
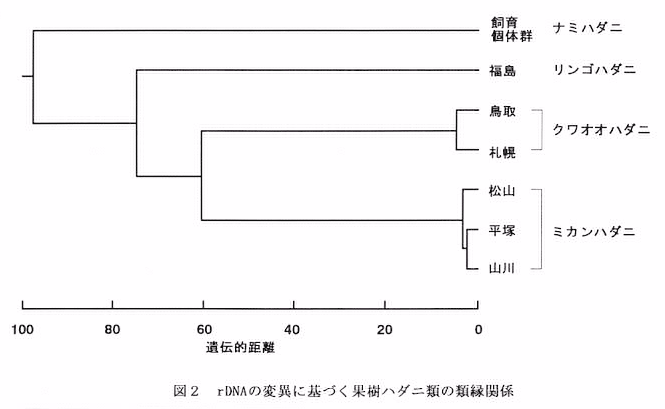
- これらの断片長の一致率から個体群間の遺伝的距離を推定し,類縁関係を検討した(図2)。その結果,ミカンハダニおよびクワオオハダニの種内変異は,種間の変異に比べてきわめて小さく,またクワオオハダニはリンゴハダニよりもミカンハダニとの類縁関係が近いことが推察された。
成果の活用面・留意点
ハダニ類の種内および種間変異の解析に寄与する。
DNAの解析法は薬剤抵抗性などの遺伝的要因を解析するための基礎技術として有用である。
具体的データ
その他
- 研究課題名:ハダニ類の種および系統間における遺伝形質の変異に関する研究
- 予算区分:平成元~4年・大型別枠研究(生態秩序)
- 研究担当者:刑部正博,坂神泰輔
- 発表論文等:RFLP analysis of ribosomal DNA in sibling species of spider
mite, genus Panonychus (Acari:Tetranychidae), Insect Molecular
Biology,vol. 3(1) 1994.
