サツマイモ二倍体近縁野生種Ipomoea trifidaのゲノム配列情報
要約
サツマイモ近縁野生種Ipomoea trifidaの固定系統Mx23Hmのゲノム解読で得られた512.9メガ塩基対のDNA配列中には、62,403箇所の遺伝子領域が存在する。ゲノム配列情報および遺伝子情報はデータベースにまとめ公開している。
- キーワード:サツマイモ、Ipomoea trifida、二倍体近縁野生種、ゲノム配列、遺伝子
- 担当:ブランド農産物開発・カンショ品種開発・利用
- 代表連絡先:q_info@ml.affrc.go.jp、FAX:096-242-7769、TEL:096-242-7682
- 研究所名:九州沖縄農業研究センター・作物開発・利用研究領域
- 分類:研究成果情報
背景・ねらい
サツマイモは世界の食用作物のうち生産量で第8位を占める重要な作物であるが、他殖性の同質六倍体であることからゲノム解析が困難で、重要形質に関する遺伝子レベルでの研究が進んでいない。サツマイモに最も近縁な野生種Ipomoea trifidaは、ゲノム解析の比較的容易な二倍体系統が存在しており、そのゲノム配列の解読は、サツマイモの遺伝的研究やDNAマーカー開発など育種にも利用出来ると考えられるため、Ipomoea trifidaのゲノム配列情報を解読する。
成果の内容・特徴
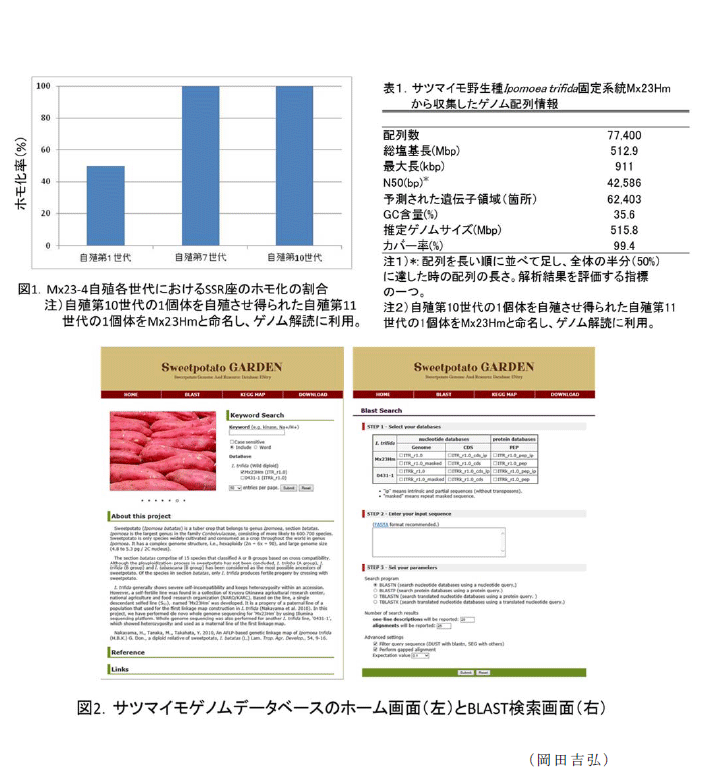
- Ipomoea trifidaのMx23Hm系統は1965年にメキシコから導入された自殖性の二倍体個体Mx23-4を第11世代まで自殖して得られた固定系統である(図1)。
- 次世代シーケンサー(Illumina社Hi-seq2000)を用いて収集したMx23Hmの配列データ(98.4 Gbp)をアセンブル(再構築)したDNA配列の総塩基長は512.9Mbpであり、K-mer分析から推定されるゲノムサイズ(515.8Mbp)の99.4%の領域に相当する。ScaffoldのN50サイズは42,586 bpであり、遺伝子配列の予測に十分な精度である。(表1)。
- 解読されたゲノム配列上に予測された遺伝子領域は62,403箇所である(表1)。
- ゲノム配列情報および遺伝子情報はかずさDNA研究所のホームページからデータベース公開(http://sweetpotato-garden.kazusa.or.jp/)されており、遺伝子配列のキーワード検索及びBLAST検索が可能である(図2)
成果の活用面・留意点
- データベースに公開された情報は、国内および世界各国のサツマイモ試験研究機関で利用出来る。
- ゲノム解読により病虫害抵抗性、デンプン合成経路等のサツマイモ生産における重要形質の選抜マーカー開発が加速され、六倍体であるサツマイモ育種へ利用できる。
- 本成果は、かずさDNA研究所との共同研究による成果である。
具体的データ
その他
- 中課題名:高品質・高付加価値で省力栽培適性に優れたカンショの開発
- 中課題整理番号:320b0
- 予算区分:交付金
- 研究期間:2003~2014年度
- 研究担当者:岡田吉弘、平川英樹(かずさDNA研)、白澤健太(かずさDNA研)、中山博貴、田淵宏朗、吉永優、高畑康浩、田中勝、田畑哲之(かずさDNA研)、磯部祥子(かずさDNA研)
- 発表論文等:Hirakawa H. et al. (2015) DNA Research 22:171-179
