牛C群ロタウイルスの全遺伝子解析
要約
世界で初めて牛C群ロタウイルスの全遺伝子配列を解明したところ、国内で流行している牛C群ロタウイルスは類似しており、ヒトおよび豚C群ロタウイルスとは分類上区別される。
- キーワード:牛C群ロタウイルス、全遺伝子解析、系統樹解析、分類
- 担当:家畜疾病防除・ウイルス感染症
- 代表連絡先:電話 029-838-7708(情報広報課)
- 研究所名:動物衛生研究所・ウイルス・疫学研究領域
- 分類:研究成果情報
背景・ねらい
C群ロタウイルスは世界中に広く蔓延しており、ヒトおよび牛や豚等の動物において重症性下痢を引き起こす人獣共通感染症病原体の一つである。本ウイルスは培養細胞での増殖・維持が困難であるため、それらの遺伝学性状や起源について不明な点が多く残されている。ヒトおよび豚C群ロタウイルスでは一部の株において全遺伝子が解読されているが、牛C群ロタウイルスでは未解明のままである。そこで、牛C群ロタウイルスの中で唯一培養細胞での増殖・維持が可能であるShintoku株および2010年に野外より採取されたToyama株について全遺伝子の塩基配列を解読し、比較・解析を行うことで本ウイルスにおける遺伝的多様性を調べるとともに、これらの遺伝子情報に既知の他動物由来C群ロタウイルスの遺伝子情報を加えて、全遺伝子を対象に系統樹解析を実施し、遺伝学的根拠に基づいて分類する。
成果の内容・特徴
- 牛C群ロタウイルスのShintoku株およびToyama株について全遺伝子の塩基配列を解読した。また両株間における各遺伝子の相同性は極めて高く、同一動物種内における遺伝的多様性はヒトC群ロタウイルスと同様にほとんどない。
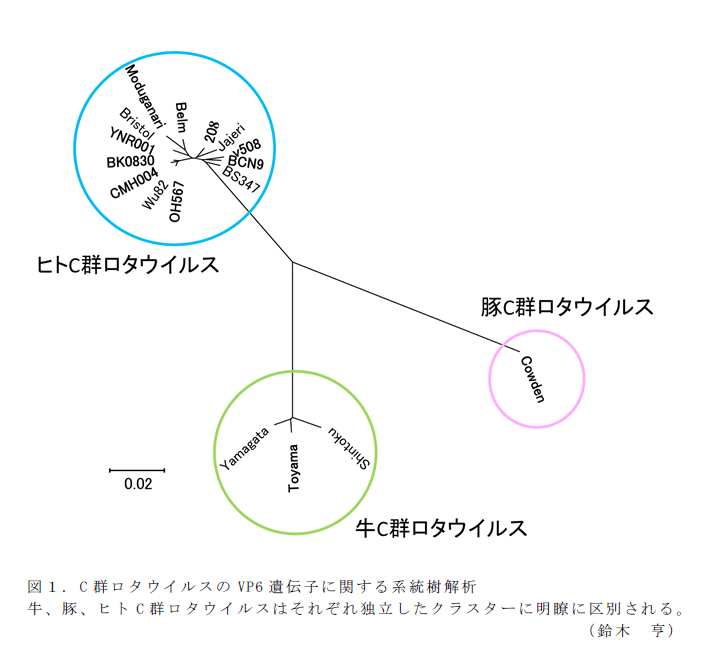
- 既知の他動物由来C群ロタウイルスの遺伝子情報に、今回新たに解読した牛C群ロタウイルスの遺伝子情報を加えて、全遺伝子を対象に系統樹解析を実施した結果、すべての遺伝子において由来動物種毎に明確に区別される。図1に既知の遺伝子情報量が多いVP6遺伝子の系統樹解析結果を示す。
- ウイルスの複製あるいは宿主免疫回避に関与するNSP1、NSP2やVP1に関して、A群ロタウイルス株間で共通して保存されている蛋白質の機能に重要な役割を果たす特異的な配列が、C群ロタウイルス株間においても高度に保存されている。そのため、これらの蛋白質はロタウイルスに共通の機能を果たしている可能性が考えられる。
成果の活用面・留意点
- 培養細胞での増殖・維持が可能である牛C群ロタウイルスShintoku株の全遺伝子情報が明らかになったことに伴い、ウイルスの細胞親和性や増殖性に関するメカニズム解明が進展することが期待される。
- 今回判明した牛C群ロタウイルスの全遺伝子情報と他動物由来C群ロタウイルス、あるいはC群ロタウイルスと同属であるA群またはB群ロタウイルスの当該遺伝子との間で塩基配列を比較・解析することで、本ウイルスの進化学的位置づけが明らかになる。
- C群ロタウイルスの全遺伝子に関する分類は、新たな変異ウイルス出現への監視に役立つと共に、本ウイルスを含めたロタウイルスの分類整備に大きく貢献する。
具体的データ
その他
- 中課題名:ウイルス感染症の発症機構の解明と防御技術の確立
- 中課題整理番号:170a1
- 予算区分:厚労科研費
- 研究期間:2010~2012 年度
- 研究担当者:鈴木 亨、相馬順一(全農)、宮崎綾子、恒光 裕
- 発表論文等:Soma J. et al. (2013) J. Gen. Virol. 94:128-135
