イネ完全長cDNAクローンの収集とゲノム配列へのマッピング、機能解析にむけたアノーテーション
-
[要約]
-
日本晴イネ由来の各種組織、ストレス処理等をおこなった組織より、完全長cDNAライブラリーを作製し、32,127個の代表クローンの全長塩基配列を決定した。ゲノム配列にマッピングした結果、20,698の転写単位に由来する遺伝子がcDNAとして得られた。これらの塩基配列情報等をデータベースとして公開した。
農業生物資源研究所・分子遺伝研究グループ・遺伝子発現研究チーム
[連 絡 先]029−838−7007
[分 類]知的貢献
[キーワード]イネゲノム、完全長cDNAクローン、マッピング、遺伝子構造、遺伝子機能
-
[背景・ねらい]
-
イネゲノム上にコードされる遺伝子の構造を正確に知るには、完全長cDNAクローンを収集し、その塩基配列を明らかにするのが最良の方法である。さらに、完全長cDNAクローンはマイクロアレイのプローブとして網羅的遺伝子発現解析や、ゲノム上のプロモーター領域の解析に、あるいはタンパク質を産生させプロテオーム的な解析に用いる等、非常に有用なゲノムツールとなる。イネ完全長cDNAプロジェクトは、当初3万を超えるユニークなクローンを収集と全長塩基配列の決定を目指した。
-
[成果の内容・特徴]
- 世界にも例を見ない3万件を越えるcDNAクローンを完全長cDNAの形で収集した。さらに塩基配列を全長に渡って極めて高精度(99.99%)で決定した。
- 日本晴イネゲノム配列に対してマッピングを行った結果、収集されたcDNAクローンはゲノム上の2万を越える遺伝子座から読まれた産物であった(図1)。
- データベースの遺伝子の塩基配列と、相同性検索を行った結果、74.5%のクローンが既登録の配列と相同性を持ち、残りの25%強が新規のクローンであった。
- ゲノム配列解読が終了しているシロイヌナズナの遺伝子(約28,000種)と比較を行うと、確保されたイネ遺伝子の約64%が両植物間で共通であり、残りがイネに特異的であった。
- データベース検索結果やシロイヌナズナ遺伝子との相同性から完全長cDNAクローンの機能分類を試みた結果、半数は機能未知、4分の1が代謝関連等であった(図2)。
- 塩基配列や相同性検索結果等のデータはインターネットから公開した。
KOME : Knowledge-based Oryza Molecular biological Encyclopedia
http://cdna01.dna.affrc.go.jp/cDNA/
- クローンに関しては増殖後、イネゲノムリソースセンターから配布できるように体制を確立した。http://www.rgrc.dna.affrc.go.jp/index.html.en
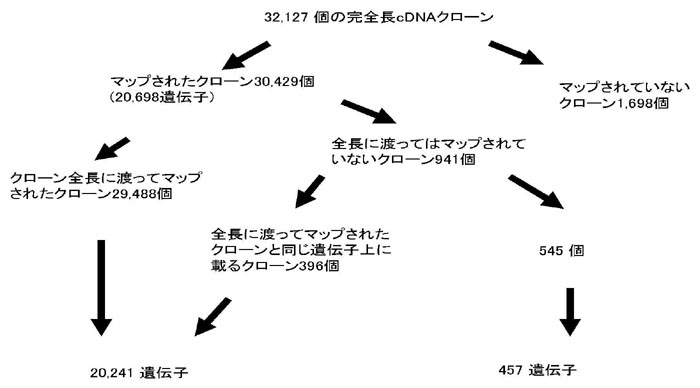
図1 |
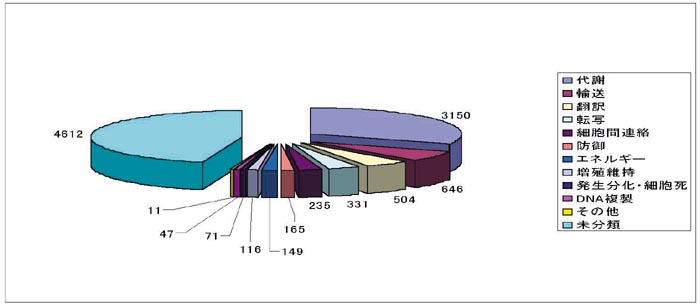
図2 |
-
[成果の活用上の留意点・波及効果・今後の展望等]
- イネ遺伝子の機能解析に極めて重要な研究リソースとなる。
- 3万2千クローンはゲノム遺伝子2万個強に相当するが、その他に端読みだけのクローンを18万保持しており、その解析も鋭意進めている。
- 完全長cDNA配列をベースとしたマイクロアレイシステムも既に製品化されており、今後はマイクロアレイを介した遺伝子発現解析の進展が期待されている。
- クローンの構造を詳細に調べることにより、オルタナティブスプライシング、アンチセンスRNA, transスプライシング等、興味深い現象が見出されており、今後の解析の進展が期待される。
[その他]
研究課題名 :イネ完全長cDNAクローンフェイズ1(32K)クローンの解析
とデータベース化
予算区分 :(イネ完全長)
中期計画課題コード:A121-1
研究期間 :99〜03年度
研究担当者 :菊池尚志,佐藤浩二,永田俊文,川頭信之,土井考爾,岸本直己,矢崎潤史,
石川雅弘,山田仁美,堀田勇ほか
(株)日立ソフトエンジニアリング,国際科学振興財団,理化学研究所
発表論文等 :1)Kikuchi S, et al.(2003)Collection, mapping, and annotation
of over 28,000 cDNA clones from japonica rice. Science,
301 : 376-379
2)菊池尚志,岸本直己,佐藤浩二,永田俊文,川頭信之,矢崎潤史,石川雅弘,
土井考爾,河合純,林崎良英,大友泰裕,松原謙一,村上和雄,植物の全長
cDNAおよびその利用. 特願2002-203269
目次へ戻る

