マイクロチップ電気泳動装置を用いた脱窒菌の菌相解析
- [要約]
- 微生物遺伝子のRsa I,Hha I,Hae IIIの制限酵素切断パターンによる分類結果と塩基配列を基にした分類結果には高い相関があり、マイクロチップ電気泳動装置で得たデータを用いることにより塩基配列の相同性検索を反映した脱窒菌の分類が可能となる。本法で家畜スラリー連用圃場のPseudomonas属脱窒菌分離割合の増加、大腸菌群に属する脱窒菌が確認される。
- [キーワード]
- マイクロチップ電気泳動装置、大腸菌群、Pseudomonas属
- [担当]
- 九州沖縄農研・環境資源研究部・土壌微生物研究室
[連絡先]096-242-1150
[区分]九州沖縄農業・生産環境(土壌肥料)
[分類]科学・参考
-
[背景・ねらい]
-
これまでに当センターで開発してきた遺伝子の類縁性検索システムによる類縁性解析結果と塩基配列の相同性検索結果との関係を検討し、分離した脱窒菌(102株)の16SrDNAをマイクロチップ電気泳動装置と本システムで類縁性解析を行い、結果を2%アガロースゲル電気泳動装置と本システム、16SrDNAの塩基配列に基づく類縁性検索結果と比較し、解析精度・分類精度・適応性の検討を行い、家畜スラリー投与の脱窒菌相への影響を明らかにする。
-
[成果の内容・特徴]
-
- 塩基配列データの非加重結合法(UPGMA)後の類似度と、計算値切断長を用いたUPGMA後の類似度の相関係数の平均値は用いる制限酵素の数(AluI,HhaI,HaeIII,RsaI,ScrF1)と共に上昇してゆき(表1)、計算上は塩基配列の相同性検索を反映した結果が得られる。
- 2%の低融点アガロースゲル電気泳動を用いた場合、5種類の制限酵素(RsaI,HhaI,HaeIII,AluI,ScrF1)を用いても切断片間の分離能が低く詳細な分類ができない菌群が生じるが、マイクロチップ電気泳動装置を用いることにより、3種類の制限酵素(RsaI,HhaI,HaeIII)で塩基配列に基づく類縁性結果と同じ分類・同定が可能となる(表2)。
- 最確値法の陽性試験管から分離した脱窒菌では、家畜スラリー投与量に伴ってβプロテオバクテリア(Burkholderia属,Ralstonia属)に属する脱窒菌の分離割合が減少し、代わりにγプロテオバクテリアに属する脱窒菌の割合が増加する(図1)。
- γプロテオバクテリアに属する細菌の内、P.stutzeriはいずれの土壌からも高い頻度で分離され、P.putida及び腸内細菌科に属する脱窒菌の分離割合が家畜スラリー投与量に伴い増加し、E.coli/Salmonella属の大腸菌群も分離される(図1、表2)。家畜スラリーの投与の影響を受けた隣接区でも60t投与区と同様な傾向を示し、大腸菌群は家畜スラリーから持ち込まれたと考えられた。
-
[成果の活用面・留意点]
-
-
本法は多数の分離菌を基にした簡易な菌相解析手法として活用できる。
-
微生物的安全性指標から有機質資材投入量を制限する際の基礎資料として活用できる。
-
システムによる分類・同定法の精度は切断長測定の精度に依存するので10%以上の測定誤差がある場合には異なった菌群が検索されてくることもある。
- 大腸菌群16S rDNAのHae III切断では166bp、161bp、156bpの切断片が生じるが、マイクロチップ電気泳動装置でもこの内2本が重なる。非特異的増幅副生物やマイナーコピー遺伝子に由来するノイズをすべて拾うためルーチン分析には1遺伝子に由来する切断片を選択的に選抜するプログラムが必要となる。
-
[具体的データ]
-
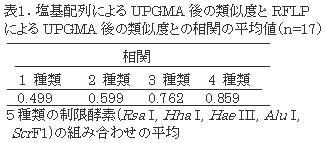
表1 塩基配列によるUPGMA後の類似度とRFLPによるUPGMA後の類似度との相関の平均値(n=17)

表2 分離脱窒菌(0t区、12t区、60t区)の16SrDNARFLP(Hae III,Rsa I)による分類
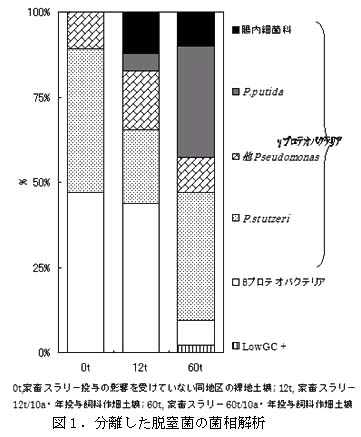
図1 分離した脱窒菌の菌相解析
-
[その他]
-
研究課題名:遺伝子の制限酵素切断長多型を利用した効率的な微生物の類縁性解析手法の開発
課題ID:07-06-02-02-05-03
予算区分 :運営費交付金
研究期間 :2000〜2003年度
目次へ戻る


