SNP判別PCRを利用したツマグロヨコバイ抵抗性イネの効率的選抜技術 | ||||
|
| ||||
| [要約] | ||||
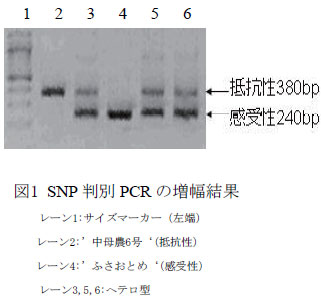
ツマグロヨコバイ抵抗性遺伝子をもつイネのDNAマーカーによる選抜には、SNP判別PCRを用いる方法が、CAPS法を用いる従来の方法より、簡便かつ低コストである。 | ||||
[キーワード]育種選抜、DNAマーカー、ツマグロヨコバイ抵抗性、SNP判別PCR |
||||
[担当]千葉農総研・生物工学部・遺伝子工学研究室 [代表連絡先]電話:043-291-9534 [区分]関東東海北陸農業・生物工学 [分類]研究・参考 |
||||
|
| ||||
| [背景・ねらい] | ||||
交配系統のツマグロヨコバイ抵抗性遺伝子座の検出は、これまではCAPS法を用いて行っていたが、この方法ではPCR反応液を精製せずに制限酵素処理をすると結果が不安定になることがあった。また、CAPS法によるDNA多型の検出は、PCRによる増幅と制限酵素による切断の2つの手順をふむため作業が煩雑になり、さらに、ランニングコストが高いなどの欠点がある。このため、育種選抜のように多数のサンプルを処理する場合にはさらに簡便、低コストな方法の開発が望まれる。そこで、DNA多型の検出を簡便かつ正確、低コストで行える技術開発を行う。 | ||||
[成果の内容・特徴] | ||||
| ||||
[成果の活用面・留意点] | ||||
| ||||
|
| ||||
| ||||
[その他] | ||||
研究課題名:イネのツマグロヨコバイ抵抗性育種に供するDNAマーカーのSNPsマーカー化 | ||||
|
| ||||
| 目次へ戻る | ||||