ダイズBACライブラリーを用いた簡便な遺伝子の単離法
[要約]
ダイズゲノムの5.9倍に相当する2種類のBACライブラリーとスクリーニング用三次元プールを構築した。スクリーニング用三次元プールを用いることにより、目的とする遺伝子配列を含むBACクローンをPCRによって迅速かつ簡便に単離することができる。
[キーワード]ダイズ(「ミスズダイズ」)、連鎖地図、BAC、TAC、遺伝子単離
[担当]北海道農研・地域基盤研究部・冷害生理研究室
[連絡先]電話011-857-9312
[区分]北海道農業・基盤研究、作物・生物工学
[分類]科学・参考
[背景・ねらい]
ダイズにおいてSSRマーカーやRFLPマーカーなど多数の分子マーカーが開発されているが、その多くは特定の品種、系統間でなければ多型を示さない。しかし、ゲノム上には塩基配列の挿入や欠失、一塩基多型(SNPs)などの未利用の多型が数多く存在する。そこで、分子マーカーや特定遺伝子を含む領域のゲノム構造を解析するため、ダイズ連鎖地図の作製に用いられている品種「ミスズダイズ」から巨大ゲノム断片を調製し、BAC(バクテリア人工染色体,Bacterialartificialchromosome)ライブラリーを構築する。さらに、分子マーカーや特定遺伝子を含むBACクローンを迅速かつ簡便に単離するシステムを開発する。
[成果の内容・特徴]
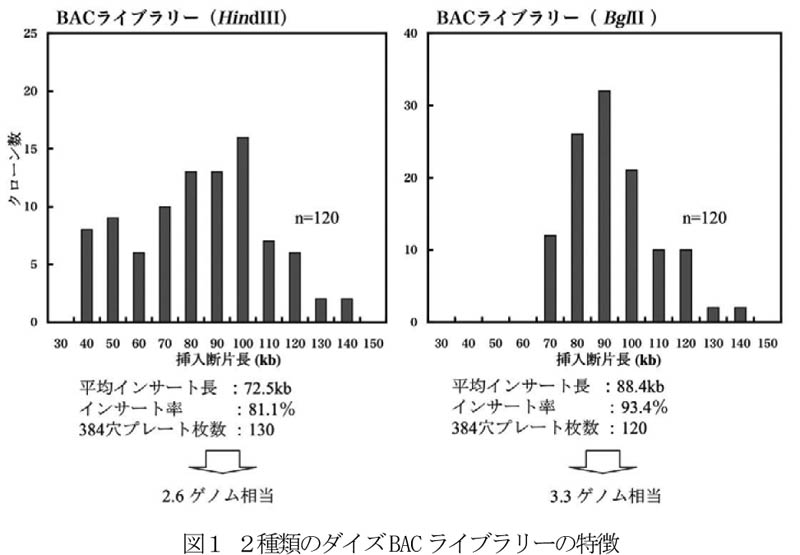
- BACライブラリー(HindIII)の総計は約2.9Gb、BACライブラリー(BglII)の総計は約3.8Gbであり、合計でダイズゲノムDNAの5.9倍に相当する(図1)。
- これらのライブラリーから分子マーカーや特定遺伝子の塩基配列を含むBACクローンを単離する手順を図2に示す。
手順A 384個の異なるBACクローンを含む保存用プレート(384穴プレート)毎に精製した一次元プールを調製し、分子マーカーや特定遺伝子を増幅するPCRプライマーによって、目的とする塩基配列を含む一次元プール(保存用プレート)を特定する。例では3枚目のプレート(#3)。
手順B 次に縦あるいは横方向に溝を切った縦・横プレート(ジェノティックス社製)に大腸菌培養培地(LB培地)を満たし、保存用プレートのBACクローンを移し、増殖させる。縦プレート(二次元プール)については横方向24列および横プレート(三次元プール)については縦方向16行について、同じPCRプライマーによって、目的とする塩基配列を含む行と列を決定し、BACクローンを特定する。例ではF行目9列目であることから、#3F09が目的の塩基配列を含んだBACクローンである。
手順C BACクローンの塩基配列を解析し、目的とする塩基配列が含まれていることを確認する。
- スクリーニング用三次元プールをすでに作製し、保存してあるため、短時間(数日間)で目的とする塩基配列を含むBACクローンを単離することができる。
[成果の活用面・留意点]
- BACライブラリーの構築にはTAC(pYLTAC7)ベクターを用いたので、アグロバクテリウム法によって挿入断片を導入することが可能である。
- BACライブラリーは2種類の異なる制限酵素による部分消化によって構築したが、まれにライブラリーに含まれないゲノム領域が存在する可能性がある。
- これらBACライブラリーの使用については、問い合わせにより対応する。
[具体的データ]
[その他]
研究課題名:ダイズのBACライブラリーの作出
課題ID:04-08-02-*-21-04
予算区分:DNAマーカー
研究期間:2002?2004年度
研究担当者:石本政男、山本直樹(東北大院理学)、柴田大輔(かずさDNA研)
目次へ戻る