公募課題
2012年6月26日更新
遺伝子発現情報のプロファイリング (略称:発現プロファイル、RTR)
概要
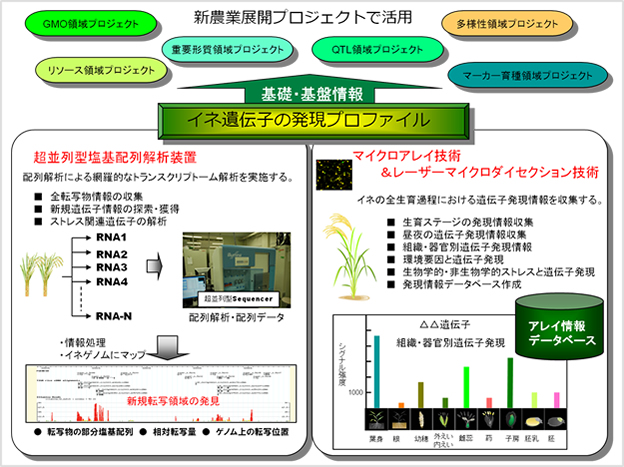
イネのゲノム配列が完全解読された今日、ゲノム上にコードされている遺伝子の同定と機能解析は重要な研究領域である。本プロジェクトでは、最新のマイクロアレイ技術や超並列型塩基配列解析装置(次世代シーケンサー)を用いた転写産物の解析手法により、イネの全転写産物の情報獲得を目指す。また、得られた個々の遺伝子の発現情報をデータベース化し、遺伝子機能解明の基盤となる情報リソースを整備する。
研究目的
- イネゲノム研究のさらなる加速化・深化を図るため、イネの全転写産物情報の獲得と各遺伝子の発現情報を収集する。
- 超並列型塩基配列解析装置を用いて全転写産物の網羅的解析を行ない、新規遺伝子の発見・獲得及び転写産物量を推定する。
- マイクロアレイ手法により、イネの全生育過程における遺伝子発現プロファイルを作成する。
達成目標
- 超並列型塩基配列解析装置を用いたイネ全転写産物の解析と新規な機能遺伝子の獲得を目指す。
- イネの全生育ステージ(種子の発芽から登熟・老化まで)における各組織・器官の遺伝子発現情報のプロファイリングを行なう。
- 生物学的ストレス(細菌、カビ)条件下及び非生物学的ストレス(温度、湿度等の環境ストレス)条件下における遺伝子発現情報のプロファイリングを行なう。
- 胚及び胚乳組織の遺伝子発現プロファイルと収量性に関わる遺伝子群の探索を行なう。
- 5年間で2,000件以上の遺伝子発現情報の収集を行ない、遺伝子発現情報データベース(発現アトラスデータベース)を作成する。
研究内容
- 超並列型塩基配列解析装置を用いたトランスクリプトーム高次解析系(配列解読系、情報解析系)を構築し、栽培種・野生種イネの全転写産物の解析を行なう。
- 自然条件下(圃場)で育成したイネ日本晴の全生育ステージにおける各組織・器官のサンプリングを行ない、マイクロアレイ解析により発育ステージ別発現プロファイル及び組織・器官別発現プロファイルを行なう。
- 病原菌(いもち菌や白葉枯病菌等)感染時におけるイネの遺伝子発現情報を収集するとともに、さらにレーザーマイクロダイセクションを使った細胞レベルでの発現情報収集に取り組む。
- 温度、湿度、照度等の環境要因を詳細にモニターした条件下でイネの発現情報を収集する。同じ環境要因で発現制御される遺伝子群や環境の影響を受けにくい遺伝子群の同定を行ない、その発現に寄与するシス配列を推定する。
- マイクロアレイ解析によって得られる発現情報をデータベース化し、情報リソースとして整備する。
↓図をクリックすると、新しいウインドウで、大きな図が開きます
 |
実施課題一覧(〜平成24年度)
↓課題番号をクリックすると、各実施課題の研究成果を見ることができます
| 課題番号 | 実施課題名 | 課題責任者 所属機関 |
|---|---|---|
| RTR0001 | イネのトランスクリプトーム解析 | 農業生物資源研究所 |
| RTR0002 | イネの生育過程における遺伝子発現アトラス作成 | 農業生物資源研究所 |
| RTR0003 | イネと病原菌等との相互作用における遺伝子発現解析 | 農業生物資源研究所 |
| RTR0004 | 自然環境下での遺伝子発現変動のプロファイリング | 農業生物資源研究所 |
| RTR0005 | イネの登熟制御に関わる遺伝子発現解析と分子機構解明 (平成22年度終了) | 作物研究所 |
| RTR0006 | 初期生活環における遺伝子発現プロファイルとその情報利用 | 東京大学 |







